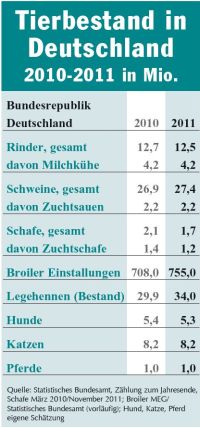
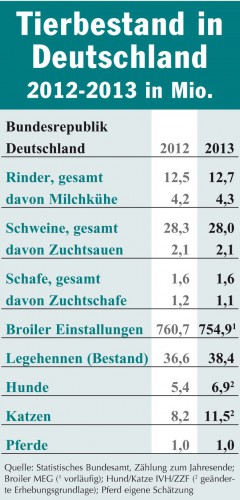
Krankenhäuser verteilen resistente Enterokokken in der Umwelt
Stockholm / Uppsala / Solna (aho) – Schwedische Krankenhäuser sind eine bedeutende Quelle von antibiotikaresistenten Enterokokken. Dieses Fazit ziehen schwedische Wissenschaftler aus ihren untersuchungen, deren Resultate sie im Fachjournal „Environmental Microbiology“ veröffentlich haben. Sie konnten einen bestimmten Ampicillin – und Ciprofloxacin-resitenten Stamm des Bakteriums Enterococcus faecium, der zur Zeit der Untersuchung in verschiedenen schwedischen Krankenhäusern vorherrschte, in verschiedenen Bereichen der Umwelt nachweisen. So waren die Keime in jeder zweiten Probe (50%) von Krankenhausabwässern nachweisbar. In kommunalen unbehandelten Abwässern waren 17% und in behandelten Abwässern 28% der Proben mit den antibiotikaresistenten Keimen belastet. Selbst in Oberflächengewässern wurden sie in jeder dritten Probe fündig (35%). Sehr selten war der untersuchte Enterokokkenstamm in Stuhlproben von gesunden Kindern (0.8%) und bei Tieren (0,2% / Schlachttiere: Rinder, Schweine, Geflügel) nachweisbar.
Die Wissenschaftler folgern, dass antibiotikaresitene Enterokokken im großem Maße von Krankenhäusern über Abwässer in die Umwelt gelangen und dort wieder Menschen infizieren. Dies werden vermutlich noch durch den Antibiotikaeinsatz in Krankenhäusern befördert. Schlachttieren zur Lebensmittelproduktion spielten bei diesem Vorgang in Schweden eine zu vernachlässigende Rolle (1).
Alle Enterokokken-Arten leben gewöhnlich im Darm von Mensch und warmblütigen Tieren, wo sie einen großen Teil der Darmflora ausmachen. Infektionen mit Enterokokken betreffen meist Patienten mit einem geschwächten Immunsystem. Innerhalb der letzten Jahre ist eine Zunahme der Infektionen mit Enterococcus spp., insbesondere E. faecalis und E. faecium, in der Humanmedizin zu beobachten. Enterokokken nehmen zur Zeit den zweiten bis dritten Platz unter den bakteriellen Erregern von Infektionen in Krankenhäusern ein. Ein besonderes Problem bei Enterokokken ist dabei ihr breites Spektrum an natürlichen und erworbenen Resistenzen gegen zahlreiche Antibiotika. Immer häufiger werden Enterokokken-Stämme bei Tieren und beim Menschen gefunden, die resistent gegen therapeutisch bedeutsame Antibiotika sind. Mehrfach resistente Enterokokken-Stämme werden deshalb als ein ernsthaftes Risiko betrachtet. Nicht geklärt ist bisher die Frage, inwieweit das Auftreten Antibiotika resistenter Enterokokken- Stämme beim Menschen allein durch den Einsatz von Antibiotika in der Humanmedizin hervorgerufen oder auch durch Resistenzen bei Tieren beeinflusst wird.
Ende der 90iger Jahre sorgten aufsehenerregende Berichte aus dem Robert Koch Institut (Außenstelle Wernigerode) (2) über die Selektion von vankomycinresistenten Darmbakterien bei Schweinen und Hühnern durch den antibiotischen Leistungsförderer Avotan (Wirkstoff Avoparcin) für Furore. Anscheinend wurden diese resistenten Darmbakterien (Enterokokken) dann über die Nahrungskette auf den Menschen übertragen. Dort siedelten sich diese resistenten Keime in der Darmflora des Menschen. Im Einzelfall konnten dann diese Keime zu schwer beherrschbaren Infektionen führen, die auch nicht mit dem Reserveantibiotikum Vankomycin behandelbar waren. Das entstehende Mediengetöse und eine heftige politische Auseinandersetzung führte dann in der Folgezeit zu einem weitgehenden Verbot antibiotischer Leistungsförderer in der EU.
Wissenschaftler der „University of British Columbia“ in kanadischen Vancouver und der „University of California“ im amerikanischen Berkeley kommen in der Fachzeitschrift „Emerging Infectious Diseases“ zu einer völlig anderen Erklärung. Sie hatten sich die Mühe gemacht und einmal näher hingeschaut. Dabei war ihren aufgefallen, dass als Leistungsförderer ein sogenanntes Feed Grade eingesetzt wurde, in dem neben dem Antibiotikum noch Reste des Bakteriums enthalten war, welches zur Produktion des Antibiotikums Avoparcin genutzt wurde. Das Bakterium trägt die Bezeichnung „Amycolatopsis orientalis C329.2“ und hat ganz natürlich die für die Vankomycinresistenz verantwortlichen Resistenzgene „vanH“, „vanA“ und „vanX“. Und genau diese Resistenzgene konnten die Wissenschaftler im Feed Grade nachweisen. Nach Meinung der Wissenschaftler haben Darmbakterien, bei der Verdauung diese Resistenzgene aufgenommen und in ihr Erbgut eingebaut. So gelangten sie zu einer Resistenz, bei der das Antibiotikum als solches keine ursächliche Rolle spielte. Bei den allermeisten Leistungsförderen wurde in der Vergangenheit keine sogenannten Feed Grades eingesetzt. Es wurde mit Reinsubstanz und einem neutralen Trägerstoff gearbeitet.
(1) Iversen A.; Kühn I.; Rahman M.; Franklin A.; Burman L.G.; Olsson-Liljequist B; Torell E.; Möllby R. Evidence fortransmissionbetween humans and the environment of a nosocomial strain of Enterococcus faecium Environmental Microbiology January 2004, vol. 6, no. 1, pp. 55-59(5)
(2) Witte W. Medical consequences of antibiotic use in agriculture. Science. 1998 Feb 13;279(5353):996-7.
(3) Lu K, Asano R, Davies J. Antimicrobial resistance gene delivery in animal feeds. Emerg Infect Dis Vol. 10, No. 4 April 2004
 ®
®






 Topic®-Emulsionspray und Gel decken ab, lösen Verkrustungen, trocknen aus und sind gegen Bakterien und Pilze konserviert. Die Topic®-Produkte neutralisieren den tierspezifischen Eigengeruch und Gerüche von Entzündungssekreten z.B. beim Zwischenschenkelekzem, Sommerekzem, Kannibalismus, Mauke, Huf- oder Klauenveränderungen. Der versorgte Bereich wird so für Insekten (Fliegen) wenig attraktiv. Die Emulsion und das Gel können auch unter Verbänden und im Zwischenschenkelbereich von Kühen eingesetzt werden.
Topic®-Emulsionspray und Gel decken ab, lösen Verkrustungen, trocknen aus und sind gegen Bakterien und Pilze konserviert. Die Topic®-Produkte neutralisieren den tierspezifischen Eigengeruch und Gerüche von Entzündungssekreten z.B. beim Zwischenschenkelekzem, Sommerekzem, Kannibalismus, Mauke, Huf- oder Klauenveränderungen. Der versorgte Bereich wird so für Insekten (Fliegen) wenig attraktiv. Die Emulsion und das Gel können auch unter Verbänden und im Zwischenschenkelbereich von Kühen eingesetzt werden.