Erneut belegt: Antibiotikaresistente Keime von Menschen und Vieh nicht identisch
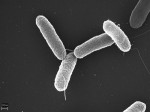 [Salmonella typhimurium DT104; Foto: V. Brinkmann, Max Planck Institut, Berlin] Hinxton (aho) – Der verbreitete Ansicht, nach der multiresistente Salmonellen beim Menschen ihren Ursprung im einer exzessiven Anwendung von Antibiotika bei Tieren in der Landwirtschaft haben, widersprechen jetzt Wissenschaftler vom Wellcome Trust Sanger Institute in Hinxton bei Cambridge. Die Experten hatten das Erbgut von Salmonellen des Typs Salmonella typhimurium DT104 analysiert, die in den letzten 22 Jahren in Schottland isoliert worden waren. Hierzu gehörten 142 Isolate von Patienten und 120 Isolate von Tieren, zumeist Rindern. Zusätzlich wurden 111 Isolate von Mensch und Tieren aus anderen Ländern in die Erbgutanalyse einbezogen.
[Salmonella typhimurium DT104; Foto: V. Brinkmann, Max Planck Institut, Berlin] Hinxton (aho) – Der verbreitete Ansicht, nach der multiresistente Salmonellen beim Menschen ihren Ursprung im einer exzessiven Anwendung von Antibiotika bei Tieren in der Landwirtschaft haben, widersprechen jetzt Wissenschaftler vom Wellcome Trust Sanger Institute in Hinxton bei Cambridge. Die Experten hatten das Erbgut von Salmonellen des Typs Salmonella typhimurium DT104 analysiert, die in den letzten 22 Jahren in Schottland isoliert worden waren. Hierzu gehörten 142 Isolate von Patienten und 120 Isolate von Tieren, zumeist Rindern. Zusätzlich wurden 111 Isolate von Mensch und Tieren aus anderen Ländern in die Erbgutanalyse einbezogen.
Salmonella typhimurium DT104 ist gegen die Antibiotika Ampicillin, Chloramphenicol, Streptomycin, Sulfonamide und Tetrazyklin resistent und bereitet deshalb bei der Behandlung Schwierigkeiten. Da der Erreger sowohl beim Menschen als auch bei Tieren auftritt, war spekuliert worden, dass durch den Einsatz von Antibiotika in der Tierhaltung antibiotikaresistente Salmonellen entstünden und über infiziertes Fleisch zum Menschen gelangten.
Das Ergebnis der Untersuchungen überraschte die Wissenschaftler. Die Salmonellen von Menschen und Tieren hatten keinen einheitlichen Stammbaum. Die bei den Tieren isolierten Bakterien unterschieden sich deutlich von denen, die beim Menschen Krankheiten ausgelöst hatten. Ein gemeinsamer Ursprung der multiresistenten Bakterien sei nicht erkennbar. (1)
Vergleichbare Ergebnisse legten Humanmediziner des „Universitair Medisch Centrum“ (UMC) in Utrecht unlängst anlässlich eines Kongresses in Berlin (23nd European Congress of Clinical Microbiology and Infectious Diseases (23nd ECCMID)) (2) vor. Sie hatten sich mit der Frage beschäftigt, ob die ESBL-Coli-Keime beim Menschen mit denen beim Geflügel identisch sind. Wie die Wissenschaftler mittels einer sogenannten Sequenzanalyse des Erbgutes feststellen konnten, waren die antibiotikaresistenten Keime nicht identisch.
(1) Mather, A. E. and Reid, S. W. J. and Maskell, D. J. and Parkhill, J. and Fookes, M. C. and Harris, S. R. and Brown, D. J. and Coia, J. E. and Mulvey, M. R. and Gilmour, M. W. and Petrovska, L. and de Pinna, E. and Kuroda, M. and Akiba, M. and Izumiya, H. and Connor, T. R. and Suchard, M. A. and Lemey, P. and Mellor, D. J. and Haydon, D. T. and Thomson, N. R.,
Distinguishable Epidemics of Multidrug-Resistant Salmonella Typhimurium DT104 in Different Hosts
Science Express 2013 DOI: 10.1126/science.1240578
(2) M. de Been, J. Scharringa, Y. Du, J. Hu, Z. Liu, Y. Lei, Z. Cen, J.W.T. Cohen Stuart, A. Fluit, M.A. Leverstein-van Hall, M.J.M. Bonten, R. Willems, W. van Schaik
Whole genome sequence-based epidemiological analysis of ESBL-producing Escherichia coli.
23nd European Congress of Clinical Microbiology and Infectious Diseases (23nd ECCMID)
27 – 30 April 2013, Berlin, Germany.
 ®
®







 Topic®-Emulsionspray und Gel decken ab, lösen Verkrustungen, trocknen aus und sind gegen Bakterien und Pilze konserviert. Die Topic®-Produkte neutralisieren den tierspezifischen Eigengeruch und Gerüche von Entzündungssekreten z.B. beim Zwischenschenkelekzem, Sommerekzem, Kannibalismus, Mauke, Huf- oder Klauenveränderungen. Der versorgte Bereich wird so für Insekten (Fliegen) wenig attraktiv. Die Emulsion und das Gel können auch unter Verbänden und im Zwischenschenkelbereich von Kühen eingesetzt werden.
Topic®-Emulsionspray und Gel decken ab, lösen Verkrustungen, trocknen aus und sind gegen Bakterien und Pilze konserviert. Die Topic®-Produkte neutralisieren den tierspezifischen Eigengeruch und Gerüche von Entzündungssekreten z.B. beim Zwischenschenkelekzem, Sommerekzem, Kannibalismus, Mauke, Huf- oder Klauenveränderungen. Der versorgte Bereich wird so für Insekten (Fliegen) wenig attraktiv. Die Emulsion und das Gel können auch unter Verbänden und im Zwischenschenkelbereich von Kühen eingesetzt werden.




One Comment, Comment or Ping
Otto
Resistenzen
Sep 14th, 2013
Reply to “Erneut belegt: Antibiotikaresistente Keime von Menschen und Vieh nicht identisch”